環境DNAは簡単に言うと生き物から離脱したDNAのことで、このDNAが例えば水中や土壌など環境中にしばらくDNAの状態で存在するので環境DNAと呼ばれています。最近このDNAは様々な場所から検出される事が分かってきており、例えば昆虫が食べた植物の葉からその昆虫のDNAが検出されたり、動物に食われた野菜からその動物のDNAが検出されたり、足跡からも検出されることもあります。さらには空気中や数百年前の海底地層にも存在することが分かってきています。このように色々な場所から検出されることから、環境DNA関係の研究が現在盛んに行われており、産業界への導入も近い状態になってきました。そんな中これから始めてみたいと言う方も多いと思います。
そこでこのサイトでは環境DNAの事は知っているが実際の検出や測定は初心者という方や、環境DNA学会から出しているマニュアル(以降学会マニュアルもしくは単にマニュアルと呼ぶ)の方法は知っているがもっと簡単な方法を知りたい方などのためにいくつかの環境DNAの測定方法を分かりやすく説明します。技術情報も参考にしながらお読みください。
(青下線文字は詳細ページに飛びます)
環境DNA測定には次のような工程があります。
- 採水;川や池から100mLから1000mLの水を汲む
- フィルタリング;採水した水をフィルタに通し、環境DNAを含む粒子を集める
- 抽出;集めた粒子からDNAを抽出し、PCR増幅できる状態にする
- PCR増幅試薬の準備
- PCRによるDNA検出;目的のDNAがあるかどうかを判定する
各工程では学会マニュアル以外にもいくつかの選択可能な方法があり、全貌は次の図のようになります。
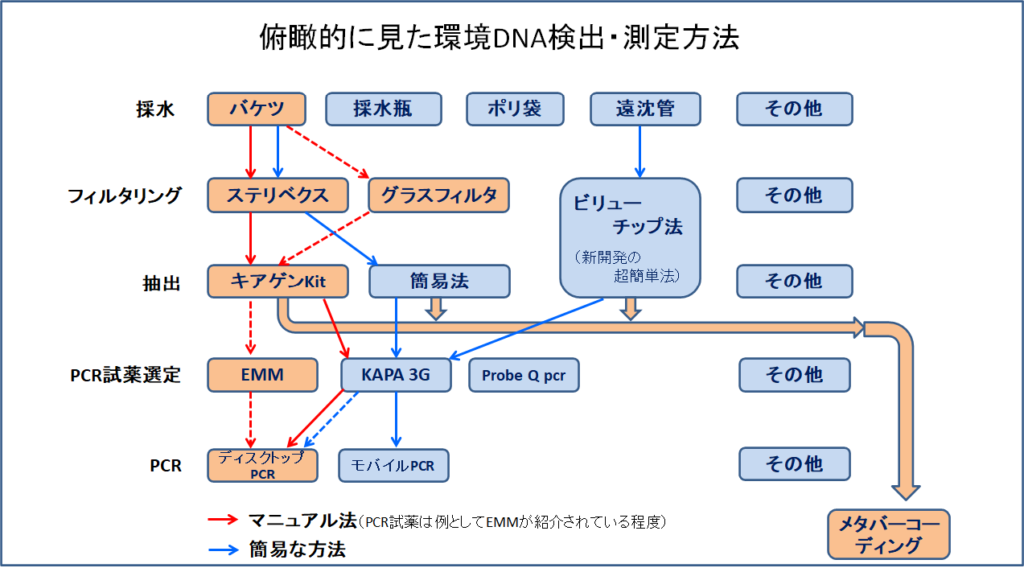
ここでは「特定の生物がいるかどうか、およびどの程度いるか」の調査・測定をするためのリアルタイムPCR法を用いたいくつかの方法について、リストを示しそのメリット・デメリットを示すと共に作業手順を説明します。各工程における手順はリンクを貼っていますのでタイトル部分をクリックして下さい。
「どのような生物がいるか」を調査するためのメタバーコーディングの方法も抽出までは同じですので参考になると思います。
目次
1.採水
2.フィルタリング
3.抽出
4.PCR試薬準備
5.PCR増幅
採水方法
どの方法にも共通しますが、偏りのない測定を行うために一測定分の水サンプルを得る時、何度か採水しこれを混ぜてその地点の平均的な水にする必要があります。混ぜる方法はフィルタリング時に混ぜても構いません。
1-1.バケツを使用する方法(マニュアル記載法)
100均ショップでも売っているバケツにロープをつけて投げ込む方法です。最も単純な方法ですが、欠点として表面の水しか取れない事、橋の上等水面から高い場所からは採水しにくいことがあります。高い場所からの採水にはこちらに記載の方法が便利です。
1-2.採水瓶を使用する方法
水面に手が届くようでしたらよく洗ったペットボトルを逆さに沈め水面下でボトルの口を上に向ければ採水できます。ただし手からのコンタミを避けるよう注意が必要です。
水面に手が届かない場所からでは蓋のついた採水瓶を沈めて水中で蓋を開ける必要があります。このような採水瓶はネットで3万円程度で販売しています(「採水瓶による採水方法」参照)。ただこれだと2本のロープが必要となり、橋の上等から採水しようとするとロープが絡んで結構使いにくそうです。
どちらもその地点周辺の平均的な水を得るためには複数回採水して別のボトルに移し替える必要があります。
1-3.その他の方法(ビリュー式採水方法)
50mLのシリンジを治具につけて水面下に沈めて採水する方法です。
長所はコンタミの心配や次亜塩素酸のすすぎ残しの心配がほとんどないことや採水後にそのままステリベクスで濾過する事ができることです。また水面の浮遊物を避けて採水することができます。
短所としては1回の採水量は50mLに限られますが、もともと何度も採水して混合することを考えると短所とは言えないかもしれません。

フィルタリング方法
2-1.ステリべクスを使用する方法(マニュアル記載法)
50mLシリンジにサンプル水を吸い上げ、ステリべクスと言うカートリッジフィルタ(構造はこちら)につなげて濾過する動作を繰り返します。所定量の濾過が終わったら空気を入れたシリンジでステリべクスの中の水を排出しそれぞれの蓋で閉めて完了です。

ろ過は500mL~1000mL行うことになりますので、シリンジ動作は10~20回行います。ただし池や川の中流以下ですと200mLも濾過できないこともしばしばあります。(ろ過量の目安はこちら)
メリットは簡単なこととコンタミの心配が少ないことで、採水した場所でフィルタリングするには非常に便利です。欠点はランニングコストがかかる事くらいだと思います。
注意事項
濾過後すぐに抽出を行わない場合は冷凍するかマニュアルにあるようにRNAlaterを充填するようにしましょう。と言いながら私はRNAlaterはほとんど使いません。理由はどうもRNAlaterを使用するとデータがばらつくような気がする故です。そのため濾過したらすぐに抽出するか冷凍しています。
2-2.グラスフィルタを使用する方法(マニュアル記載法)
長所はランニングコストが安いことですが、短所は必要な機材が多く初期投資が必要なこととそれなりの場所が必要な事、さらにコンタミの心配が多い事です。手順は現在作成中です。

2-3.ビリュー式濾過法
これは独自に開発中ですが、下写真のような微細流路を用いてフィルタリング&抽出をほぼ同時に行うことができる方法です。実験の結果では、ステリベクスを使った方法と比較して1/20の濾過量で同等以上の検出感度が得られています。従ってこれまで何本もの1Lボトルを分析場所に冷蔵で送って大変な費用と労力を要していたものが、50mL(遠沈管1本分)で済みます。しかも抽出をほぼ同時に簡単に行うことができます。

抽出方法
3-1-1.キアゲンの抽出キットを使用する方法(学会マニュアル記載法=AL法)
キアゲン社のDneasy Blood and Tissue kitを使用した方法です。
手順はかなり複雑で初めてだとかなり面倒だという印象を持つと思いますが、慣れれば1.5時間程度で抽出できます。
学会のマニュアルに従うとかなり高価な遠心機や恒温槽・ローターなどいろいろ初期に機材をそろえる必要がありまが、これまでの経験ではもう少し安くなる方法はありそうです。

この方法のメリットは
- DNAの抽出効率が良いので環境DNA測定の感度が良くなる事、(ただし最近これに疑問符がついています)
- 世の中で広く認知された方法であるため信頼されている事、
- 抽出後のサンプルは不純物が少ないので安定して保管できる事です。
一方でデメリットは
- 手順が多く時間がかかる事、
- スキルが必要なこと、
- 必要な機材が多く初期投資が高いこと、
- さらにサンプル中の不純物によって抽出効率が10倍以上変動することがあります。
3-1-2.キアゲンの抽出キットを使用する方法(ATL法)
上の学会マニュアル記載法を少し改良した方法です。わずかな改良により収率が2倍程度良くなると共に夾雑物による抽出効率の変動が大きく改善するようでお勧めです。手順はマニュアル法と類似で約1.5時間程度で完了します。
3-2.簡易(HS)抽出方法(ゴーフォトン社開発)
使用する処理液はA液とB液の2液のみ(下写真参照)であり、手順は非常にシンプルで処理時間は5分以内です。

この方法のメリットは
- 手順が少なく所要時間が短い事、
- スキルがあまり必要としない事、
- 必要な機材が少なく初期投資が少なくて済む事、
- 現場でもできる(電源が不要)事です。
一方でデメリットは
- 抽出後のサンプルに不純物が残りやすい故、冷凍しておかないとDNA量が減る事
- 同じ理由で場合によって夾雑物に強いPCR試薬が必要になります。
PCR試薬に「KAPA 3G」を使用した場合では検出限界はマニュアルにあるキアゲン法と変わりませんでしたので、抽出効率は変わらないと考えられます。
これから環境DNA測定を始めたいという方には好適な方法と思います
なおこの原理はHot-Shotという手法がベースになっていると思われ、勝手にHS抽出法と書かせて頂きました。興味のある方はネットで調べれば自分で試薬を安く作れるかもしれません。
3-3.Biomemeの抽出キットを使用する方法
この方法は知人が横でやっているのを見ただけですが、ピペッターも必要なく素人でも簡単に抽出ができるようです。しかし残念ながら感度を考えるとメリットを感じない結果と聞きました。検出対象やPCR試薬との相性もあったかもしれませんので今後の結果も待ちたいと思います。
3-4. ビリュー式抽出法(BC法)
フィルタリングのところでも触れましたが微細流路チップを使用してフィルタリングと抽出を同一チップ内でほぼ同時に行うことができ、採水後5分程度でろ過&抽出ができます。しかも実験では1/20のサンプル水量で同じ感度(なので感度20倍と言える?)が出ています。手順が極めて簡単なため初心者でも簡単にできます。

3-5その他
その他の抽出法として、磁気ビーズを使用した方法やフェノクロと呼ばれる方法、キアゲンの方法を若干変更した方法等があります。
磁気ビーズを使用した方法は感度・簡易度・コストからみてキアゲン法と簡易法がある中で特に使いたいというメリットは今のところ私は感じていません。フェノクロはキアゲン法よりもテクニックが必要であり、バラツキが多いため初めての方にはお勧めできない方法と思います。
DNA検出試薬の準備
PCR増幅するにあたって、どのようなPCR試薬を使ってどのPCR増幅装置を使用するかを検討しなければなりません。また試薬と装置の相性もあり、特定の装置を使用する場合、使用できない試薬があることもあります。
従来の高価なPCR装置ですと他にも使える試薬が多くあると思いますが、「簡単に測定できる」ことを中心に検討していますので、今まで検討した試薬は主に以下3試薬になります。
4-1.Environmental Master Mix2.0(=EMM)
(学会マニュアル記載試薬)
環境DNA学会が出しているマニュアルに例として記載されている試薬です。自分でテストしたことはありませんが、知人テスト結果や論文をみての個人的印象は以下です。
メリットは、
- 学会のマニュアルに記載されているので結果に対して信頼されやすい
ー方でデメリットは、
- 夾雑物が入っていると感度が悪くなる
(論文でもそのような結果が出ています) - 高速のPCRには対応できないことがあげられます。
(高速モードは使えない)
したがって比較的きれいな水質での調査において、キアゲン法等の不純物を除去できる抽出を行い、従来からあるディスクトップ型PCR装置で測定する場合においては使用できる試薬と思います。
4-2.KAPA 3G Plant
PCR反応が高速で且つ不純物(夾雑物)にも強いとされている試薬です。

メリットは、
- 短時間でPCR増幅が完了すること
- 夾雑物につよいこと
- 試薬が分かれていて調合の自由度があること
一方でデメリットは、
- 価格が比較的高いこと
- 試薬が分かれていて調合が煩雑なことがあげられます。 (調合の自由度と裏腹です)
抽出の部分で書きましたように、簡易抽出法は簡単で短時間に処理することができますが、不純物(夾雑物)が残るため、簡易抽出法を使用するには夾雑物に強いPCR試薬を使用する必要があります。この観点からこの試薬は非常に有能でしかも反応が早いので、これまでほとんどこの試薬を使用しています。
4-3.Probe q-PCR
上記のKAPA以外に使用できそうなものを探した結果、この試薬を見つけ知人と一緒に試してみました。
その結果としてメリットは、
- 短時間でPCR増幅が完了すること
- 夾雑物につよいこと
- 調合が簡単なこと
一方でデメリットは、
- 価格が若干高いこと
- 調合の自由度がないこと
使ってみてなかなかいい試薬という印象です。従来からあるディスクトップ型PCR装置だと(そこそこの夾雑物の場合)この試薬もよさそうに思います。
ただ難点を言うと簡易装置としてのモバイルPCR装置にはそのまま使用できない事です。これはディスクトップ型PCR装置用に組成を合わせ込んでいるのでモバイルPCR装置には若干モディファイが必要なことが理由です。またディスクトップ型PCR装置でも夾雑物耐性を更に上げようとした場合、酵素濃度を少し上げたいところですがそれができません。
4-4.その他のDNA検出試薬
PCRによるDNA増幅試薬は上記以外にも多く市販されておりますが上記に記載のものは使用実績があります。実際に使用する際はこれらの試薬に測定対象固有の配列を持った試薬(プライマー・プローブ)を適量混合し使用することになります。この配合は素人には難解ですが、例えばGoFoton社からはいくつかの測定対象について簡単に使用できる試薬セットが販売されていますので比較的に簡単に使用できます。
PCR増幅法(PCR装置とその使用法)
PCR法でもいくつかの方法があり、ここではリアルタイムPCRという方法の中でプローブをもちいた方法が可能な装置を紹介します。他の方法はPCR増幅の後ひと手間ふた手間かけないと結果がでないためです。
5-1.ディスクトップ型PCR装置と操作手順(マニュアル記載法)
この部類の装置は以前から多くの装置が販売されています。有名なのは例えば以下があり、これらは遺伝子分析の分野でかなりの実績もあります。
- ロシュ社 ライトサイクラー
- タカラバイオ社 ダイス
- サーモフィッシャー社 StepOne
など
これらは使い勝手には差がありそうですが、性能はどこも似たようなものと思います。ただこれらは安いものでも200万円程度しますので、これから始めようという方にはハードルが高いかも知れません。

これらの装置のメリットとしては
- 販売実績が長く安心して使用できること
- 一度に最大96検体の測定ができること
一方デメリットは
- 装置が高価なこと
- 装置が大きく場所をとること
- 操作手順が複雑であること
- PCR増幅反応時間が長いこと
これから多くのサンプルを測定したい方はこのディスクトップ型PCR装置から選べばよいかと思います。
5-2.モバイルPCR装置と操作手順
現在携帯型のPCR装置で国内で入手でき、実際に携帯して使用できるのはPicogene PCR1100(日本板硝子社)だけだと思います。
(日本板硝子社HPより)
この装置のメリットとしては
- 小型で電池駆動のためどこでも測定できる事
- 測定時間が短い事
- 操作が簡単なこと
- 装置が比較的安い事
一方デメリットは
- 1サンプルづつしか測定できない事
- 専用のチップ(反応容器)が高い事があります。
この装置は測定対象にあった増幅条件を選んでボタンを押すだけで測定ができ、15分程度で結果が出ます。携帯型は最近登場したものですが、個人的な使用実績は多いです。
気になる性能ですが、環境DNA測定に置いてディスクトップPCR装置(StepOne)と比較した結果ほとんど変わりませんでした。
5-3.その他の装置
モバイルとは言いにくいですが、車で持ち運べる程度の機動性のあるものとしては以下のものもあります
・タカラバイオ社 CronoSTARTM Portable
・クラボウ社 Model GF-Q150
・ My go-mini
など
これらPiciGene、CronoSTAR、GF-Q150をカタログベースで比較したものはこちら。
最後に
以上これまで種々の方法を記載してきましたが、他にも方法があると思い、これからも順次追加していく予定です。
ここで最初に示した俯瞰図を再度お見せしますが、この図で赤矢印で書いたマニュアル法に比べて青矢印で書いた簡易法では作業がかなり簡単になっています。

そこで気になるのが簡易法は環境DNA検出感度がどうかと言うことと思います。これについては先に示したように抽出での差もPCR装置での差もほとんど変わらないので全体として変わらないを想像できると思いますが、実際に検出全体をマニュアル法と簡易法で比較した結果もありますので後日掲載する予定です。

