1.はじめに
今回は今後環境DNA測定に役立つと思われる論文(http://doi.org/10.1002/edn3.406)の紹介になります。
環境DNA学会のマニュアルに記載されているキアゲン(Qiagen)社のDneasy Blood and Tissue kitを使用した環境DNAの抽出法は別投稿で説明していますが、色々実験していると場所や時期により抽出効率がかなり変動することが分かり悩んでいました。そこで少し改良すると抽出効率が高くなるだけでなく変動も小さくなるようなので実験内容と共に手順を紹介します。この方法をここではATL法と呼びます。使用する機材はマニュアル法と同じでこれも慣れれば1.5時間程度で終わります。
2.論文の実験内容
まずニジマスの水槽水を大量に確保し、良く混合後50mLの遠沈管に分注して冷凍したニジマススパイク水を大量に作ります。
次に相模川と霞ヶ浦の水を毎週採水し、そこにニジマススパイク水を一定の割合で混合したものを測定サンプルとしました。これを100mLづつろ過し学会法(AL法と呼ぶ)と新規提案のATL法で抽出してニジマスを対象に定量PCRを行いました。ろ過は前半(2020年;実験1)はステリベクス、後半(2021年;実験2)はグラスフィルタです。この測定を1年以上実施しています。またこのサイトで紹介しているビリューチップ(BC)法での測定も一部行っています。
相模川の結果は図-1のようになっています。
もし安定した抽出ができていればいつも一定のcopy数がでるはずですが、AL法はかなり変動していることが分かります。一方でATL法は安定し高収率で抽出できている事がわかり、BC法も簡易な方法ながら安定し比較的高収率で検出できています。ここで実験1はステリべクス、実験2はグラスフィルタのろ過です。
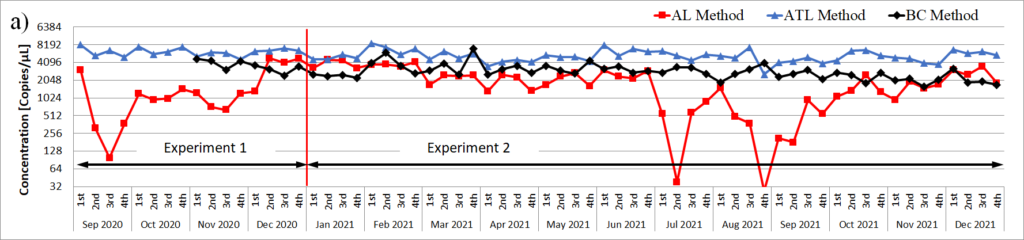
図-2が霞ヶ浦の結果です。こちらも相模川と同じような結果が出ています。
![Difference in extraction efficiency when changing lysates during fixed-point observation of environmental DNA.
At Kasumigaura]](https://biryu-kikaku.com/wp-content/uploads/2022/07/霞ヶ浦結果-1024x240.png)
論文ではスパイクしたニジマスの他に現地に生息しているのアユ(相模川)とハクレン(霞ヶ浦)でも定量PCRを行っていますが、AL法とATL法の比はニジマスと同様の傾向を示しています。つまり実生息種でも今回のニジマスの結果と同様にAL法では夾雑物により変動が大きくなる(収率が低下する)と考えられます。
ちなみにAL法での収率低下はPCR阻害による低下では無いことを別途確認しています。
3.抽出作業で準備するもの
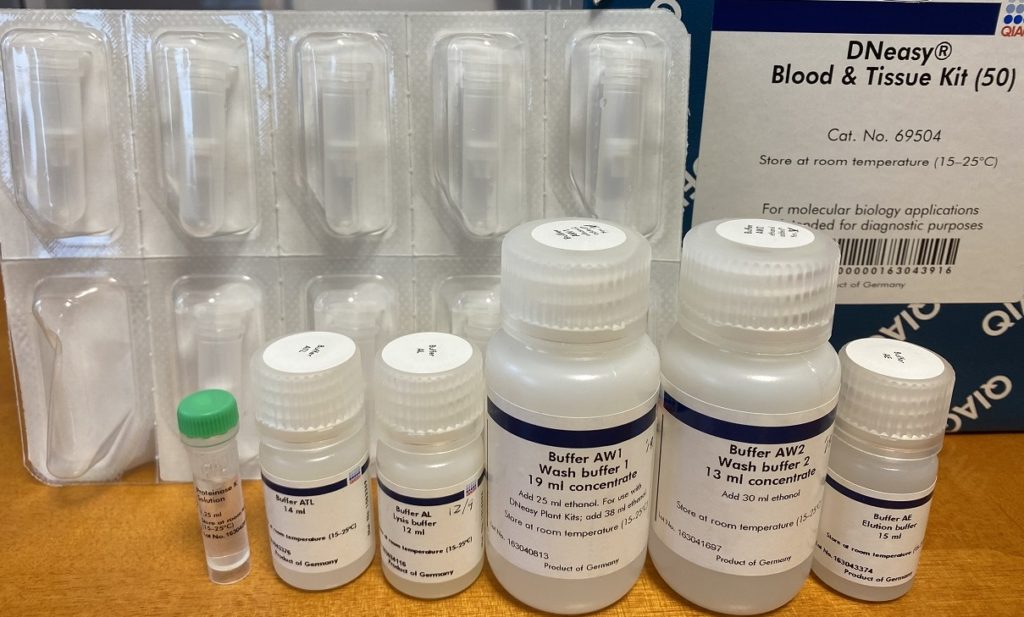
Dneasy Blood and Tissue kit内に上写真の試薬が入っています。具体的には写真左から
- Proteinase-K(Pro-K)
- Buffer-ATL(AL法=学会法では使用していない)
MSDSによるとSDS(sodium dodecyl sulphate)のようです - Buffer-AL
- Buffer-AW1
- Buffer-AW2
- Buffer-AE
となっています。ここで間違いやすいポイントとして、AW1,AW2を最初に使用する前にエタノールをまぜる必要があることを忘れないようにしましょう。
これにエタノールが必要になります。
キット内には下図のスピンカラムが入っていて(上の写真では後ろ側)、これを使用して処理をします。
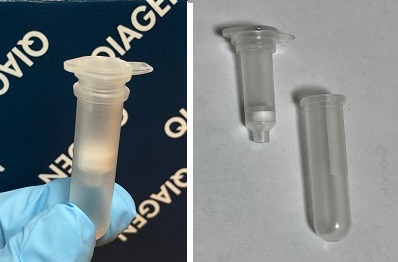
(下側をコレクションチューブと呼びます)
4.論文記載の抽出手順
ステリベクスの場合の抽出手順を次に示します。太字部分がAL法からの主な変更点です。グラスフィルタの手順は追って追加しますが、現時点では論文をお読みください。
- Buffer ATL 380 µLとProt-K 20 µL割合で処理数分混ぜたプレミックスを作製。この時同時に複数サンプルの処理をする場合は各容量の1.05倍の量を混ぜることをお勧めします。
- 上記プレミックス400uLをピペッターでステリベクスに充填
ピペッターのチップは必ず毎回交換してください - 56℃-30分処理 この時ローテーターなどを使用してステリベクスを回転させる
- 遠心してステリベクスの入口側から液を1.5mLチューブに取り出す
- このチューブにBuffer-AL400uLを入れて撹拌し56℃-10分処理
- このチューブにエタノール400uLを入れて撹拌
- スピンカラムに上記液の約半分を入れて6000g-1分で遠心
- コレクションチューブの水を捨てた後スピンカラムに残り半分を入れて6000g-1分で遠心
- コレクションチューブの水を捨てた後、Buffer-AW1を500uL入れて6000gで1分で遠心
- コレクションチューブの水を捨てた後、Buffer-AW2を500uL入れて20,000gで3分で遠心
(10,000g程度の遠心機の場合はこの工程を2回行う) - コレクションチューブを新しい1.5mLチューブに換えてカラム上部にBuffer-AEを200uL入れて1分待つ
(このAEの量については別の投稿も参照ください。濃度を上げることができます) - 6000gで1分で遠心後、1.5mLチューブに残ったものがsampleです。
学会マニュアルの手順(AL法)と比べると最初にATLを使用する部分以外ほとんど差はありませんが、実験結果が示すように効率安定性は大きく改善することから、今後はこの方法がお勧めだと思っています。
5.その他情報
幾つか論文を読んだ印象ですが、海外の論文ではATLを使用している研究者も多いようす。ただしATLを使用していてもここで書いたような詳細な手順を見つけることはできませんでした。日本ではAL法を使っている方が多いようです。またAL法とATL法の収率差を比較できるような過去の論文は見当たりませんでした。
上にも書きましたが、Buffer-ATLの(主な)組成はSDSのようで、これは代表的な陰イオン界面活性剤でタンパク質を分解する能力があります。