1.はじめに
環境DNA学会のマニュアルに記載されているキアゲン(Qiagen)社のDneasy Blood and Tissue kitを使用した環境DNAの抽出法です。慣れれば1.5時間程度で終わります。ここで紹介するのは初心者が導入しやすいようにマニュアルを少しアレンジしています。このアレンジにより感度が悪くなっていない事は確認済です。
一方でこの方法では経験上夾雑物による抽出効率の変動がかなりあるようです。そこで少し改良することにより効率がupすると共に変動が激減することが最近分かりました。この改良した手順(ATL法)や比較実験結果は別の投稿に紹介しています。
なおこれらの方法はPCR増幅を行う前のDNA抽出法として一般的なものですので、環境DNA以外のPCR測定にも応用できる方法です。(ただしその時は対象によって少しづつ条件を変える必要が出るかもしれません)

2.準備するもの
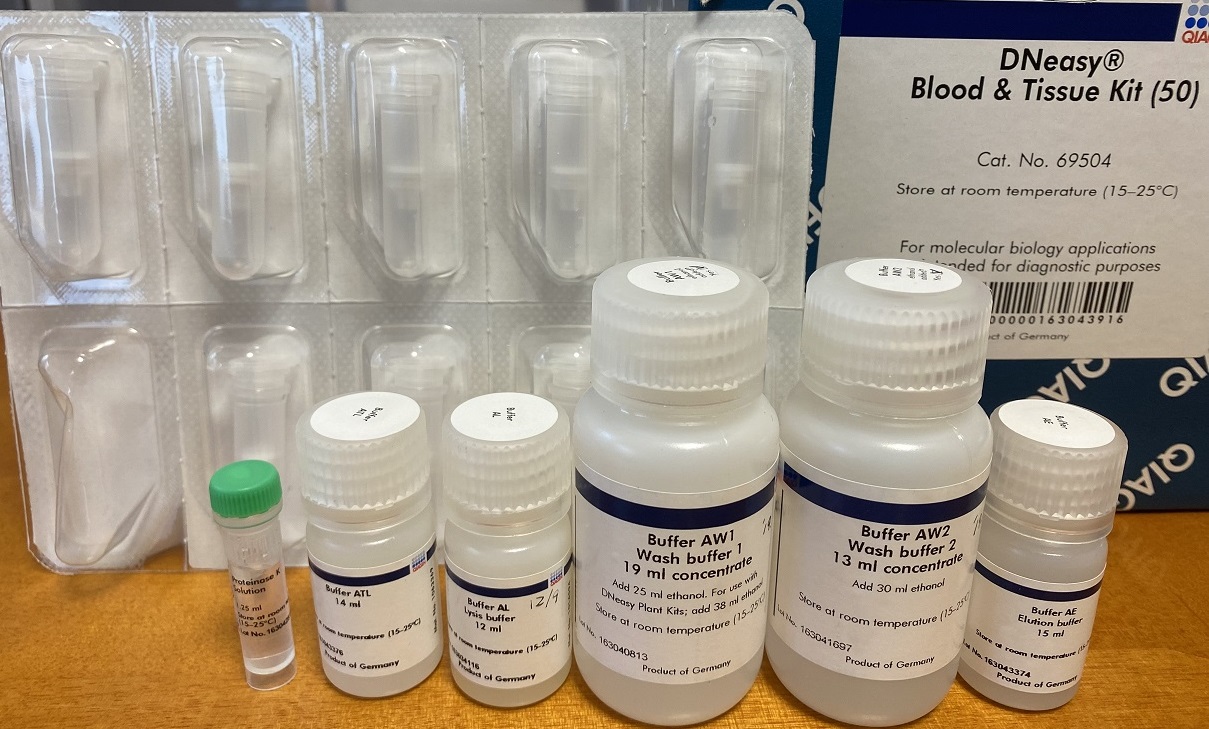
このキット内に上写真の試薬が入っています。具体的には写真左から
- Proteinase-K(Pro-K)
- Buffer-AL
- Buffer-AW1
- Buffer-AW2
- Buffer-AE
となっています。ここで間違いやすいポイントとして、AW1,AW2を最初に使用する前にエタノールをまぜる必要があることを忘れないようにしましょう。
これにエタノールとPBS(リン酸バッファー)が必要になります。
キット内には別に下図のスピンカラムが入っていて、これを使用して処理をします。
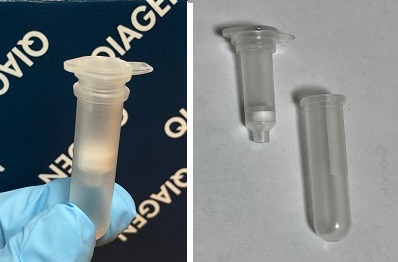
(下側をコレクションチューブと呼びます)
処理を行うにあたって必要な主な機器はこちらのリンクをご参照ください。学会マニュアルと比較した表を作成しています。
恒温槽でステリベクスを回すミニローテーターを使わない理由は「技術情報の記事」に記載していますが、お金と場所に余裕があればローテーターを使った方が無難です。
50mLのコニカルチューブが入る遠心機を使わない理由は「便利な機材の記事」 をご参照ください。
3.手順
以下の手順で抽出を行います。イラストも参照しながら読んでください。
- Pro-K;20uL、PBS;220uL、Buffer-AL;200uLの割合で処理数分混ぜたプレミックスを作製。この時同時に複数サンプルの処理をする場合は各容量の1.05倍の量を混ぜることをお勧めします。
- 上記プレミックスをピペッターでステリベクスに充填
ピペッターのチップは必ず毎回交換してください - 56℃-30分処理 この時約5分毎にステリベクスを回転させる
- 遠心してステリベクスの入口側から液を1.5mLチューブに取り出す
- このチューブにエタノール200uLを入れて撹拌
- スピンカラムに上記液を入れて6000g-1分で遠心
- コレクションチューブの水を捨てた後、Buffer-AW1を500uL入れて6000gで1分で遠心
- コレクションチューブの水を捨てた後、Buffer-AW2を500uL入れて20,000gで3分で遠心
(10,000g程度の遠心機の場合はこの工程を2回行う) - コレクションチューブを新しい1.5mLチューブに換えてカラム上部にBuffer-AEを200uL入れて1分待つ
(このAEの量については別の投稿も参照ください。濃度を上げることができます) - 6000gで1分で遠心後、1.5mLチューブに残ったものがsampleです。

次の動画も同時にご参照下さい。
なお、この手順のベースとなる反応については「技術情報の記事」をご覧頂けると覚えやすいと思います。
注)動画では学会マニュアルに従い、手順1でPro-KとBuffer-ALを最初に混ぜていますが、キアゲンのマニュアルではこの混ぜ方は避けるようにと書いています。従って気になる方はBuffer-ALは最後に入れてください。ただし私が行った実験ではどちらでも変わりませんでした。
4.その他情報
キアゲン社のDneasy Blood and Tissue kitを使用した抽出方法でも、測定対象により使用する試薬を変えるように取説には書いています。例えばkitの中にBuffer-ATLという試薬も入っており、細胞組織から抽出する場合にはこちらを使うことを推奨しています。
環境DNAのほとんどは細胞ではなくミトコンドリアとして浮遊しているDNAを測定しているようなので、Buffer-ALを使用することを学会のマニュアルで定めているものと理解しています。ただ場合によってはBuffer-ATLを使用した方が抽出効率が良くなったり、またもっと別の方法の方で抽出効率が良くなる可能性もあります。
この方法より簡単な簡易(HS)法も環境DNAの抽出に使用できます。これは手順が少なく慣れれば5分以内で抽出でき、感度も劣らないという結果が出ています。
一方でこの抽出法(学会マニュアルに準じた方法)では環境水中の不純物によって抽出効率がかなり変化することも分かってきております。これを認識したうえで使用する必要があります。
2022.6月 追記
上に書いたBuffer-ATLを使用すると学会のマニュアルの方法(ここで書いているBuffer-ALを使用する方法)よりも抽出効率が高く且つ安定していることが分かったと論文に記載されています。特に夾雑物が含まれている場合、マニュアルに従った方法ではかなり効率が落ちることがあるようです。私自身これ以外の同様のデータをいくつも持っており、常にATLの方法が同等以上の効率を示しています。それ故個人的には今後ATLを使った方法をお勧めします。手順は論文に詳しく書いていますのでご覧ください。
更に追記;ATLを使用した実験結果や手順を別投稿でupしました。