環境水を出発点とする環境DNAの検出では、夾雑物による阻害の影響を常に考えなければなりません。
環境DNA学会が発行するマニュアルや環境DNAのバイブル本(?勝手にそう思っていますが「環境DNA」共立出版発行のもの)などでもPCR阻害のことがよく書かれています。
例えばそのバイブル本の1.3.5章で「増幅曲線の立ち上がりが他のサンプルと比べて顕著に緩やかな場合などにはPCR増幅の阻害が考えられる」と書かれています。
そこで実際にどのような増幅曲線になるか測定してみました。
方法としては、
河川の水を精製工程の無い簡易法で抽出したサンプルの中から、見た目で夾雑物が多そうなものと少なそうなものを選び、この二つのサンプルをディスクトップ型PCR装置(StepOne)で川魚をターゲットに増幅してみました。この時PCR試薬にはKAPA 3G Plantを使用し、反応量は15uLでサンプルは10%にしています。
その結果を図-1に示します。ここでサンプルAは無色透明に近いもので、サンプルBはかなり色がついていて見るからに夾雑物が沢山ありそうなものでしかも沈殿物を沢山分取しました。
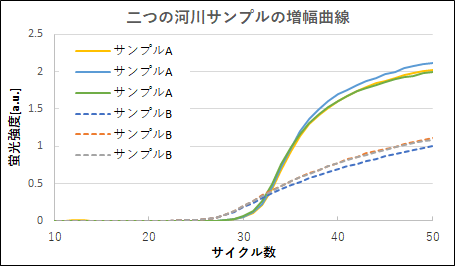
図-1より明らかなようにサンプルBの増幅曲線は顕著に緩やかなになっており、何かの阻害があったものと判断できます。
ではPCR阻害なのかそれとも抽出等が影響した阻害なのか、さらにはDNA量が少ない場合でも増幅するのかを調べるため、次のような測定をしてみました。
PCR試薬に上記二つのサンプルを10%の比率で混ぜ、それぞれに10^5から10^1copies/uLの海水魚のDNA鋳型を10%の濃度で混合。これをStepOneでN=3回繰り返しで増幅してみました。
結果を図-2に示します。


図-2からやはりサンプルBが混じると増幅曲線が緩やかになっていることがわかり、図-1の結果は増幅阻害であったことが分かるかと思います。
ただサンプルBではスパイク濃度との関係がよくわからないので、この図-2の縦軸を対数にしたものを図-3に示します。


図ー3よりどちらのサンプルも増幅初期はほぼ同じ傾きであり、同じように増幅が進み低濃度でも増幅していたことが分かります。
ちなみにこの時得られたCt値は3繰り返しの平均で次のようになっていました。
| 鋳型濃度 | サンプルA | サンプルB |
| 10^5 | 22.5 | 22.7 |
| 10^4 | 26.1 | 25.8 |
| 10^3 | 29.4 | 28.9 |
| 10^2 | 32.3 | 32.2 |
| 10^1 | 35.6 | 36.6 |
Ct値はどちらのサンプルでもほとんど差がありません。
今回使用したKAPA3GというPCR試薬は夾雑物に強いとされていますので、たまたまCt値に差が出なかったのかもしれません。しかしバイブル本でも書いていますが、Ct値だけで結果を判断をするのではなく環境DNA測定の場合特に増幅曲線も確認することが重要と思います。
ところで冒頭「環境DNA測定では夾雑物による阻害の影響を常に考えなければなりません」と書きましたが、夾雑物の影響はこのPCR阻害以外にも抽出阻害の影響も考えなければなりません。別の投稿でも書きましたが抽出方法によっては夾雑物(水質)の差で一桁以上の効率差が出ることがあります。
私の感覚ではCt値で見た場合、夾雑物による影響は増幅阻害よりも抽出阻害で主に発生していると感じています。これはフィルタリング前の環境水に一定濃度の内標をスパイクし、比較測定する実験をここしばらく重ねている関係で分かってきたものです。これまで環境水の採水地点や時期により(特に学会マニュアルにあるキアゲンキットを使用した時)増幅曲線は変わらずCt値が大きく変動するという結果を多く見てきています。
この夾雑物による阻害の影響は常に考えてておく必要があると言いましたが、阻害があるか無いかをどのように判断するかが最初の問題になります。この判断をするためにフィルタリング前の環境水に一定濃度の内標を入れて測定しチェックしておく事が必要だろうと考えています。